中科院环境生物技术重点实验室邓晔研究组与日本东京大学、四川大学、大连理工大学等单位合作,在微生物功能基因数据库开发和应用方面取得新进展,相关研究成果近日发表于Methods in Ecology and Evolution(Wang, et al. 2022),Molecular Ecology Resources (Wang, et al. 2022),Journal of hazardous Materials(Li, et al. 2022)等生态学和环境科学期刊上。
环境核酸检测技术已广泛应用于研究不同生境中的专一微生物(如新冠病毒等)或广谱微生物群体(如细菌群落等)的分布和含量状况,但这些检测技术的准确性高度依赖于PCR引物的特异性和覆盖度,而设计和评估高质量的扩增引物均需构建全面的标记基因序列数据库。同时,环境样本中广泛存在的 PCR抑制剂对痕量基因的检测也造成极大的困难。
在此,邓晔研究组开发了一个基于隐马尔可夫模型(Hidden Markov model profiles)的标记基因序列收集平台,兼顾序列的高质量和完整性。针对表征氮素转化酶的标记基因构建氮循环基因数据库NcycFunGen,包含涉及7个氮循环过程的22个标记基因,共收集60余万条蛋白质和对应的核苷酸序列及完整的物种注释信息。基于该数据库,系统评价了608条已发表的氮循环基因引物,其结果表明,大部分收集到的引物对仅能覆盖不到30%的目标序列。因此,该团队进一步通过NcycFunGen收集的高质量序列,重新设计了脲酶标记基因ureC、氨单加氧酶标记基因amoA和固氮基因nifH的简并引物对。 引物评价中,这些新设计的引物对具有更高的覆盖度和扩增效率、以及更合适的扩增子长度,可用于高通量扩增子测序。 此外,在草地尿素添加实验中,数字微滴PCR和高通量扩增子测序结果显示新设计的ureC基因和nifH基因引物对比之前发表的通用引物对具有更好的特异性和可以检测更广的物种分类范围。以上结果发表于生态学杂志Methods in Ecology and Evolution (Wang, et al., 2022)。
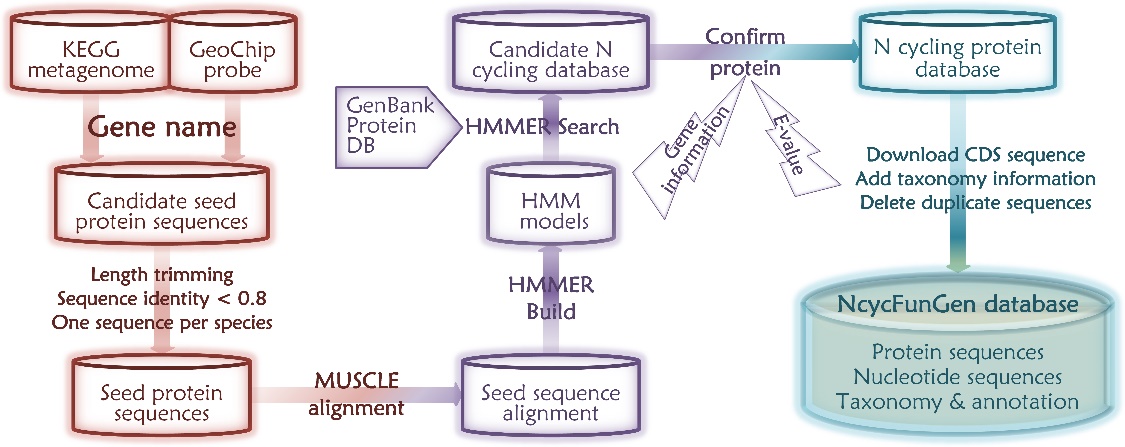
图1 数据库构建流程
与此同时,该研究团队还针对芳香族化合物降解的微生物标记基因ARHDs构建序列数据库DARHD,系统地评估了该基因的谱系多样性和底物广谱性。引物评价结果表明,目前广泛采用的引物在用于复杂的环境样本检测时还存在覆盖度低和特异性差等诸多不足,实验时需要根据样本类类型及研究目标进行特异性的引物设计 (Li, et al., Journal of Hazardous Materials, 2022)。此外,该团队还系统评估了新一代定量技术——数字微滴PCR(ddPCR)在环境样本中的表现,实验表明ddPCR在标记基因定量方面比传统定量PCR具有更好的准确性、可重复性、灵敏性和稳定性。由于该技术对PCR抑制剂不敏感,并具有定量较长目的片段的潜力,因此ddPCR在环境监测和评估中能更准确地定量特定微生物或功能类群的环境总量(Wang, et al., Molecular Ecology Resources, 2022)。
以上研究得到了国家科技部、自然科学基金和四川省科技厅有关项目支持。
原文链接
论文链接1:https://doi.org/10.1111/2041-210X.13946
论文链接2:http://www.sciencedirect.com/science/article/abs/pii/s0304389422010202
论文链接3:https://onlinelibrary.wiley.com/doi/10.1111/1755-0998.13644
中科院环境生物技术重点实验室
2022年7月27日
