中国科学院生态环境研究中心环境化学与生态毒理学国家重点实验室汪海林研究组在哺乳动物基因组DNA N6-甲基腺嘌呤研究方面取得新进展。他们发展了一套独有的、无污染的6mA分析方法,在哺乳动物细胞系中检测到的6mA并不依赖于甲基化转移酶,并发现了DNA聚合酶依赖的DNA 6mA新来源。相关工作近日以N6-methyladenine is incorporated into mammalian genome by DNA polymerase为题在Cell Research杂志上发表。
DNA N6-甲基腺嘌呤(N6-methyladenine, 6mA)是一种在原核生物中广泛存在的表观遗传修饰,但是高等真核生物中6mA的存在一直是一个难解的谜。2015年,汪海林组与陈大华组合作在Cell上发表了关于果蝇DNA 6mA修饰的研究成果。哺乳动物DNA 6mA的研究也随后开启了新的里程。但是,由于哺乳动物DNA在样品制备和分析过程中,容易受到原核生物DNA污染,而原核生物6mA 水平远高于哺乳动物的,为哺乳动物DNA 6mA的准确检测与研究带来巨大挑战。同时,研究人员也因此困惑:哺乳动物基因组DNA中是否存在复制后可遗传性的N6-腺嘌呤甲基化修饰?该问题一度引起业内广泛关注与争议。
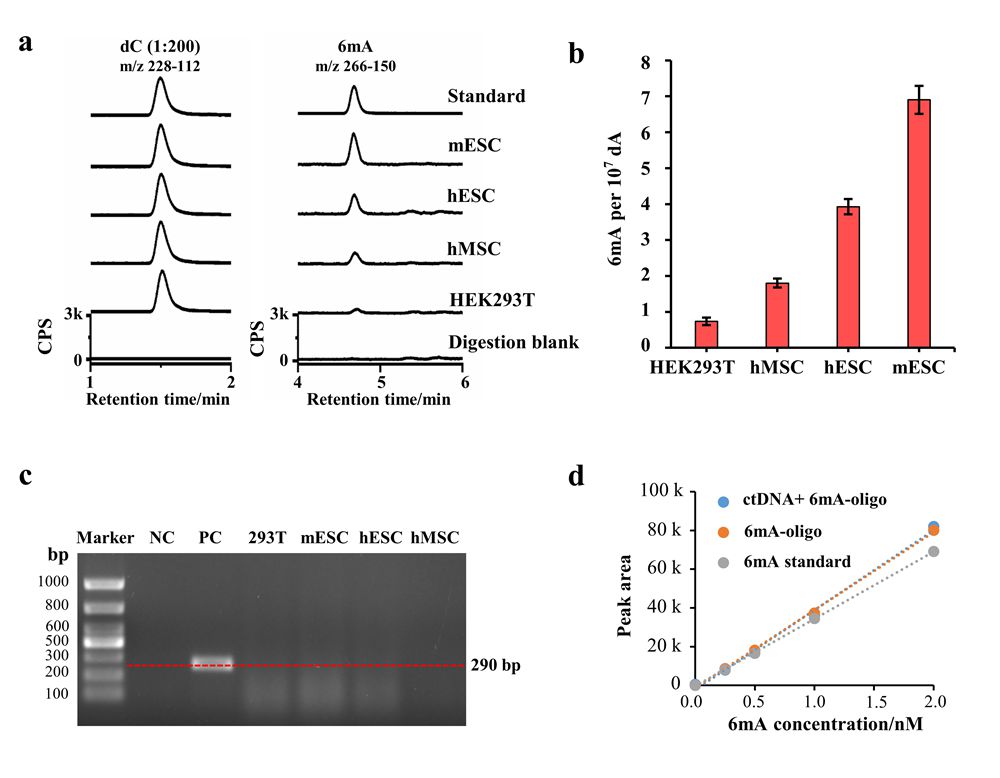
为了回答这一问题,汪海林研究组发展了一套独有的、无污染的6mA分析方法,在样品预处理、超高效液相色谱串联质谱(UHPLC-MS/MS)分析中排除可能的原核生物DNA的污染,可获得准确的6mA信号。利用这一先进的分析方法,在三种人细胞系和小鼠胚胎干细胞(mouse embryonic stem cells, mES细胞中检测到6mA(图1a, b)。研究还发现6mA可在细胞G1期累积。
为了进一步探索6mA的来源,该组采用他们研发的一种独特的稳定同位素标记方法。该法使用[15N5]-dA作为初始示踪剂处理细胞。其中,[15N5]-dA通过补救合成途径以[15N4]-dA的形式掺入基因组DNA中。理论上也可用于识别哺乳动物细胞内甲基化转移酶产生的6mA ([15N4]-6mA)。研究发现,经[15N5]-dA处理后,细胞DNA被[15N4]-dA有效标记,但未能在mESC等细胞系、不同细胞周期中检测到任何[15N4]-6mA。该研究采用了第二种有效的同位素标记试剂,即[13CD3]-L-甲硫氨酸,但仍然未检检测到任何[13CD3]-6mA。以上结果表明,在哺乳动物细胞系中检测到的6mA并不依赖于甲基化转移酶。
图1. UHPLC-MS/MS 分析哺乳动物细胞基因组DNA 6mA
他们推测细胞内6mA来源于特定DNA聚合酶的活动。据此,采用6mA核苷直接处理mES细胞,基因组DNA中可检测到剂量依赖的6mA掺入。随后,分别用同位素标记的[15N5]-6mA和RNA核苷[CD3]-m6A处理细胞进行验证,可分别检测到[15N5]-6mA和[CD3]-6mA掺入。那么mES细胞6mA是如何进入基因组的呢?研究发现,与非同步化的细胞相比,G1期细胞中DNA聚合酶X家族成员非模板依赖性的Polλ的mRNA表达增加,而Poly λ敲低则显著降低了G1期和sub G1期6mA水平。且Polλ敲除可导致[CD3]-m6A处理掺入的6mA减少。
上海生科院徐国良院士等参与了这一课题研究。
原文链接:https://www.nature.com/articles/s41422-020-0317-6
环境化学与生态毒理学国家重点实验室
2020年5月4日