在自然生境中微生物物种间如何相互作用以适应环境的极端压力?随着环境组学技术的进步,我们才有工具来探索这些微小生物之间的复杂关系。传统上,微生物共现网络被用来推测物种间的互作,但这种网络往往不能揭示微生物之间更深层的相互依存机理。近日,中国科学院环境生物技术重点实验室的邓晔团队开发完成了一种新的工作流程,该流程结合了微生物宏基因组数据的共现网络分析与微生物代谢互补潜力的预测,为探索微生物互作的潜在底层机制提供了新的视角。团队开发了一个在线分析平台(https://inap.denglab.org.cn),允许研究人员上传环境宏基因组序列,进行代谢模型预测、构建共现网络/代谢互补网络、网络参数与模块化分析,以及互补代谢物的分析与可视化等。这一工具极大地简化了微生物互作研究的复杂性,为全球科研人员提供了一个强大的分析工具。
利用这一工作流程,邓晔团队对中国云南腾冲的热泉微生物群落进行了深入分析。研究发现,共现网络和代谢互补网络识别的互作关系在自然界中是稀少的。辅酶A、氨基酸与糖类化合物及其衍生物是这些互作关系中最常见的潜在可交换物质。此外,研究还发现微生物间的协同关系主要表现为偏利共生(commensalism),而非互利共生(mutualism)。这一发现对于理解微生物如何在极端环境中生存和繁衍具有重要意义。为了验证这些发现的普适性,该团队还对基于堆肥实验的温度梯度样品进行了分析。结果表明,这一工作流程在分析的可重复性与结论的一致性上是可靠的,进一步证实了其在不同环境条件下的适用性。
相关成果分别发表在Nature Communications和iMeta上。课题组直博生彭玺为论文第一作者,邓晔研究员为通讯作者(课题组助理研究员冯凯为iMeta论文共通讯作者)。研究得到国家重点研发计划国际合作项目以及国家自然科学基金联合项目的支持。
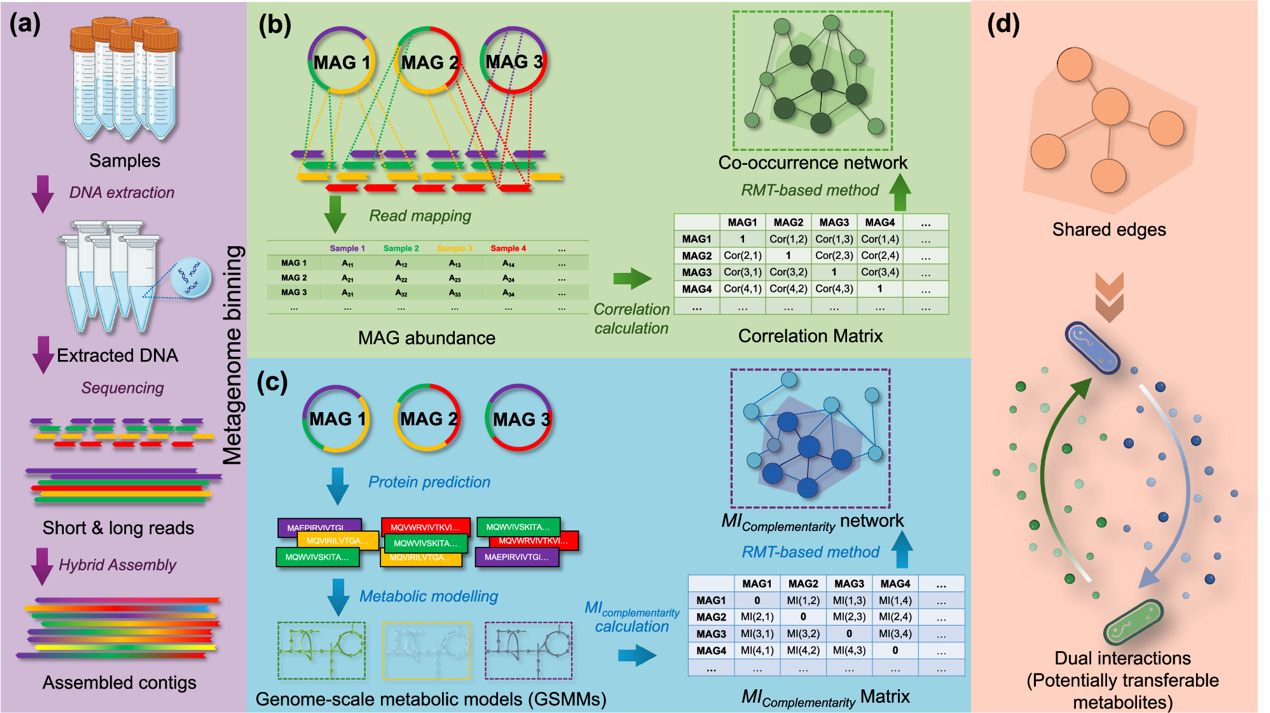
图 本研究的分析流程示意图
论文链接:
1. https://www.nature.com/articles/s41467-024-52532-x
2. https://doi.org/10.1002/imt2.235
中国科学院环境生物技术重点实验室
2024年9月19日
