2024年8月,环境化学与生态毒理学国家重点实验室彭汉勇课题组与江桂斌院士、加拿大X. Chris Le院士等团队合作,在CRISPR基因编辑工具开发方向取得新进展,研究成果以“RNA-activated CRISPR/Cas12a nanorobots operating in living cells ”为题发表在Journal of the American Chemical Society期刊上(Supplementary Cover)。

CRISPR-Cas系统作为一种革命性的基因编辑工具,因其操作简单、编辑精确、效率高等特点,已在分子生物学和基因治疗领域得到了广泛应用。CRISPR基因编辑技术中的Cas蛋白,如Cas12a和Cas13a,能够特异性靶向核酸,并激活反式切割活性,非特异性持续切割单链核酸报告基因。Cas酶的反式切割特性使得检测信号可以被放大,提高了检测的灵敏度,推动了CRISPR技术在超灵敏分析和分子诊断领域的发展。然而,目前这些Cas酶主要用于试管内分析,而在活细胞中的应用受到限制。主要原因为:一、RNA激活的Cas13a会对胞内功能性RNA进行非特异性反式切割,可能对细胞造成伤害,而Cas12a虽然仅反式切割单链DNA,但能否被胞内RNA激活尚未明确;二、活细胞内条件无法达到最佳切割活性,包括Mg2+离子、报告基因浓度等,受限于复杂的胞内递送过程。因此,亟需开发新的CRISPR工具,以满足活细胞应用需求。
环境化学与生态毒理学国家重点实验室彭汉勇团队通过研究Cas12a酶的功能,发现了Cas12a能够被RNA激活,并反式切割DNA报告基因。CRISPR/Cas12a与靶标RNA通过碱基互补配对结合,其反式切割速率随DNA报告基因的延长而增加。这一发现表明Cas12a蛋白是一种独特的“全能型”蛋白酶,能够直接靶向三种类型的核酸,包括RNA、单链DNA、双链DNA,并激活其反式切割活性。该工作还系统评价了CRISPR/Cas12a的RNA/DNA靶向能力及酶切速率,为CRISPR研究提供了定量数据。该发现为CRISPR/Cas结构与功能研究带来了新的视角,将推动病毒检测、疾病诊断、转录组调控、分子成像和基因编辑等领域的CRISPR新技术研发。
为了突破活细胞应用瓶颈,彭汉勇团队基于上述Cas12a新功能,构建了一种高度集成的RNA激活CRISPR/Cas12a纳米机器,用于活细胞中特征miRNA的成像。通过将CRISPR/Cas12a系统及其报告基因组装到纳米金球表面,实现在胞内外CRISPR体系中各组分的化学计量比始终保持不变,不受进胞量的影响。同时,在纳米金表面的有限纳米空间内,Cas12a及其底物报告基因的局部浓度较高,切割速率得到大幅提升。此外,由于Mg2+离子与核酸链上磷酸基团的相互作用,使得纳米金表面Mg2+离子的局部浓度得到提高,在低Mg2+离子浓度下Cas12a仍能保持高切割活性,从而克服了胞内低浓度Mg2+离子的限制。将CRISPR/Cas12a纳米机器与细胞进行孵育,在胞内识别特征miRNA后启动,产生持续放大的荧光信号,实现对胞内miRNA的成像。CRISPR/Cas12a纳米机器在活细胞中具有优异的RNA识别能力和胞内成像性能,融合了精准识别、高效运转与自动化操作等特性,同时展现了组件更换的设计灵活性,为开发新的CRISPR工具提供了一种个性化定制平台。
该工作发现RNA可以直接激活CRISPR/Cas12a系统的反式割活性,这一发现打破了之前仅限于DNA激活的认知,并揭示了RNA与DNA激活的Cas12a系统的基因切割行为差异。通过人工合成的CRISPR纳米机器平台,实现CRISPR系统在胞内的高效运转,有望在分子诊断、基因编辑、癌症治疗、感染性疾病和其他遗传性疾病的精准医疗中发挥重要作用,为环境健康、生物技术、医学领域的进步提供新的动力。
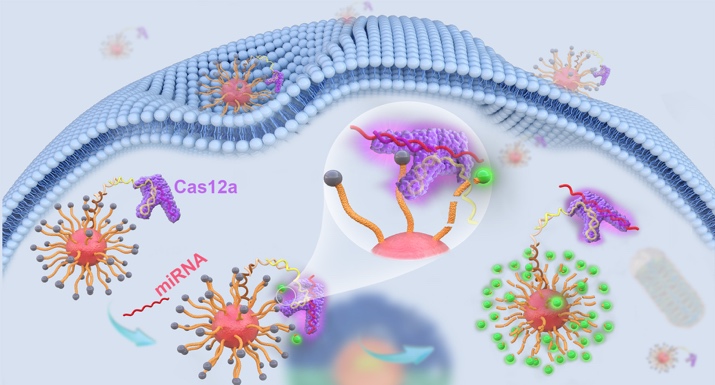
图1. RNA 激活的 CRISPR/Cas12a 纳米机器及其活细胞成像
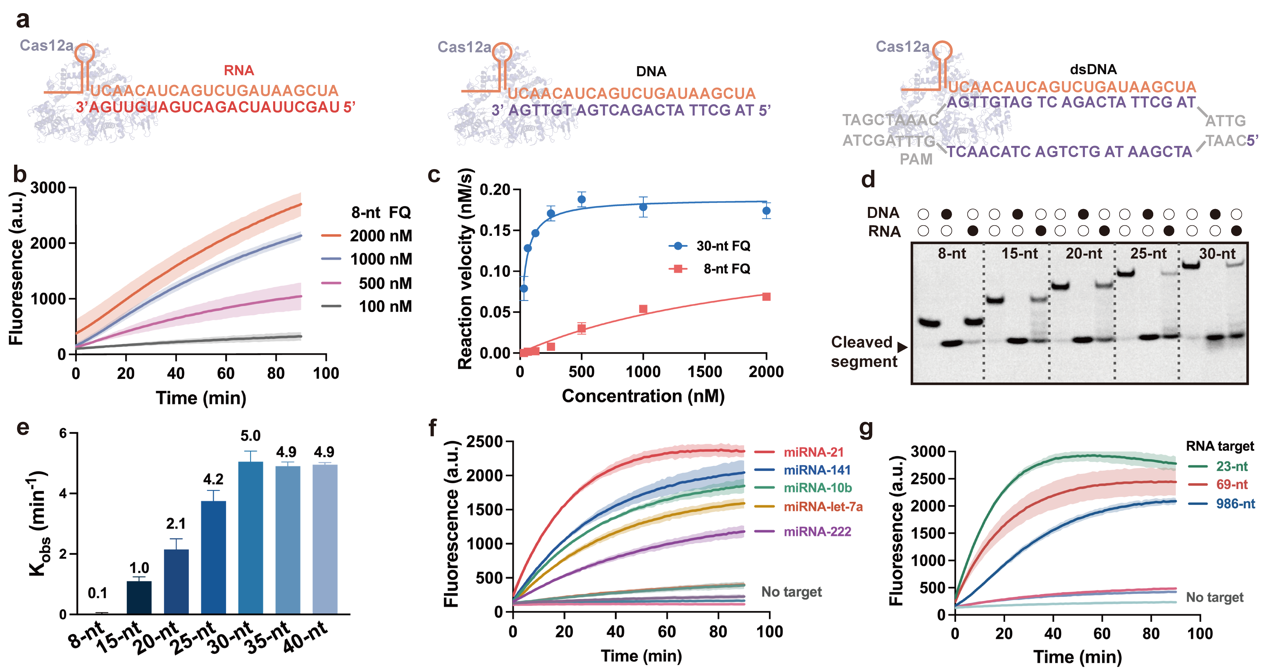
图2. RNA 激活的 CRISPR/Cas12a及其反式切割活性评价。(a)显示CRISPR/Cas12a系统的RNP与靶RNA、ssDNA或dsDNA的相互作用的示意图。(b)使用RNA激活的CRISPR/Cas12a系统通过8 nt报告基因(100、500、1000和2000 nM)的反式切割产生的荧光。(c)从溶液中miRNA激活的crRNA-Cas12a RNP对不同长度DNA报告基因的反式切割中观察到的反应速度。crRNA-Cas12a RNP的浓度为2 nM,靶miRNA的浓度为40 nM。当使用30 nt FQ reporter作为底物时,米氏常数(KM)测定为3.5 × 10- 8 M,切割速率(Kcat / KM)为2.8 × 106 M-1 s-1。当使用8 nt的报告基因作为底物时,米氏常数(KM)为2.2 × 10−6M,Kcat / KM为3.1 × 104M−1 s−1。miRNA激活的crRNA-Cas12a RNP的反式切割有利于30 nt报告基因而不是8 nt报告基因(底物)。(d)显示从8、15、20、25和30 nt底物裂解的片段的凝胶图像。CRISPR/Cas12a系统由RNA或ssDNA激活。反应30分钟后进行凝胶电泳。(e)通过RNA活化的CRISPR/Cas12a系统跨切8、15、20、25、30、35和40 nt底物的表观切割速率(kobs)。(f)在CRISPR/Cas12a系统被五种microRNA(miRNA-21、miRNA-10 b、miRNA-141、miRNA-let-7a和miRNA-222)激活后,切割30 nt报告基因的荧光。(g)在CRISPR/Cas12a系统被不同长度(23、69和986 nt)的RNA激活后,切割30 nt报告基因的荧光。
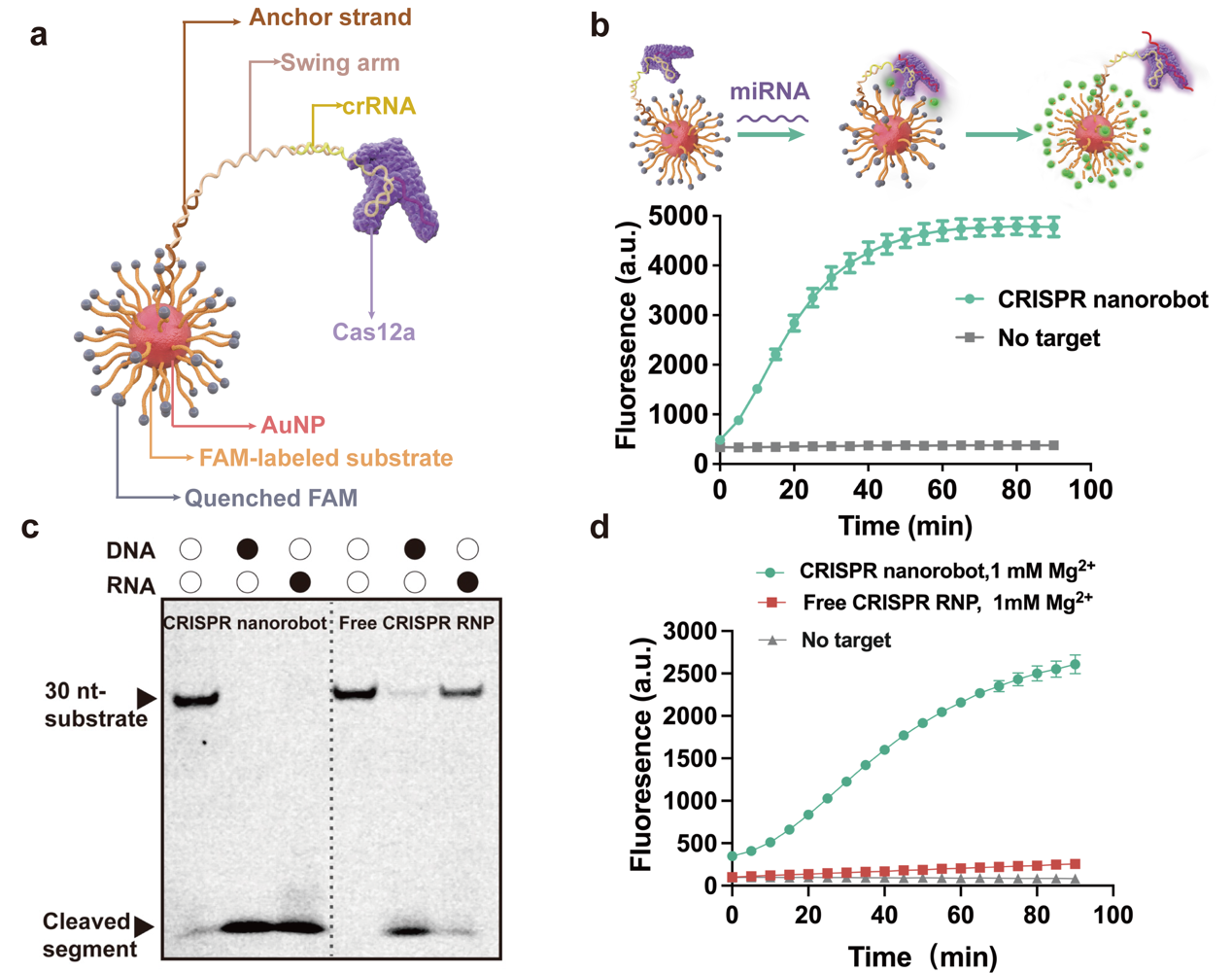
图3. CRISPR纳米机器的构建与性能评价。(a)CRISPR纳米机器由用分子构建体修饰的AuNP组成,以使CRISPR系统能够运行。ssDNA底物链的5′端被硫醇化以促进底物与AuNP的缀合。底物链的3′-末端用FAM荧光团标记,其荧光被AuNP淬灭。硫醇化的锚链也与AuNP缀合。crRNA序列被延伸以包括在一端与锚链杂交并且在另一端与延伸的crRNA杂交的摆臂。crRNA和Cas12a形成RNP复合物,该复合物组装在AuNP支架上。(b)响应于200 pM miRNA,实时监测由CRISPR纳米机器产生的荧光。(c) 凝胶电泳表征30 nt底物及其被反式切割的产物。DNA和RNA分别用于激活溶液中的CRISPR纳米机器或游离的CRISPR RNP。(d)通过CRISPR纳米机器和溶液中的游离CRISPR RNP对底物反式切割产生的荧光。Mg2+浓度为1 mM,靶miRNA-21浓度为500 pM。
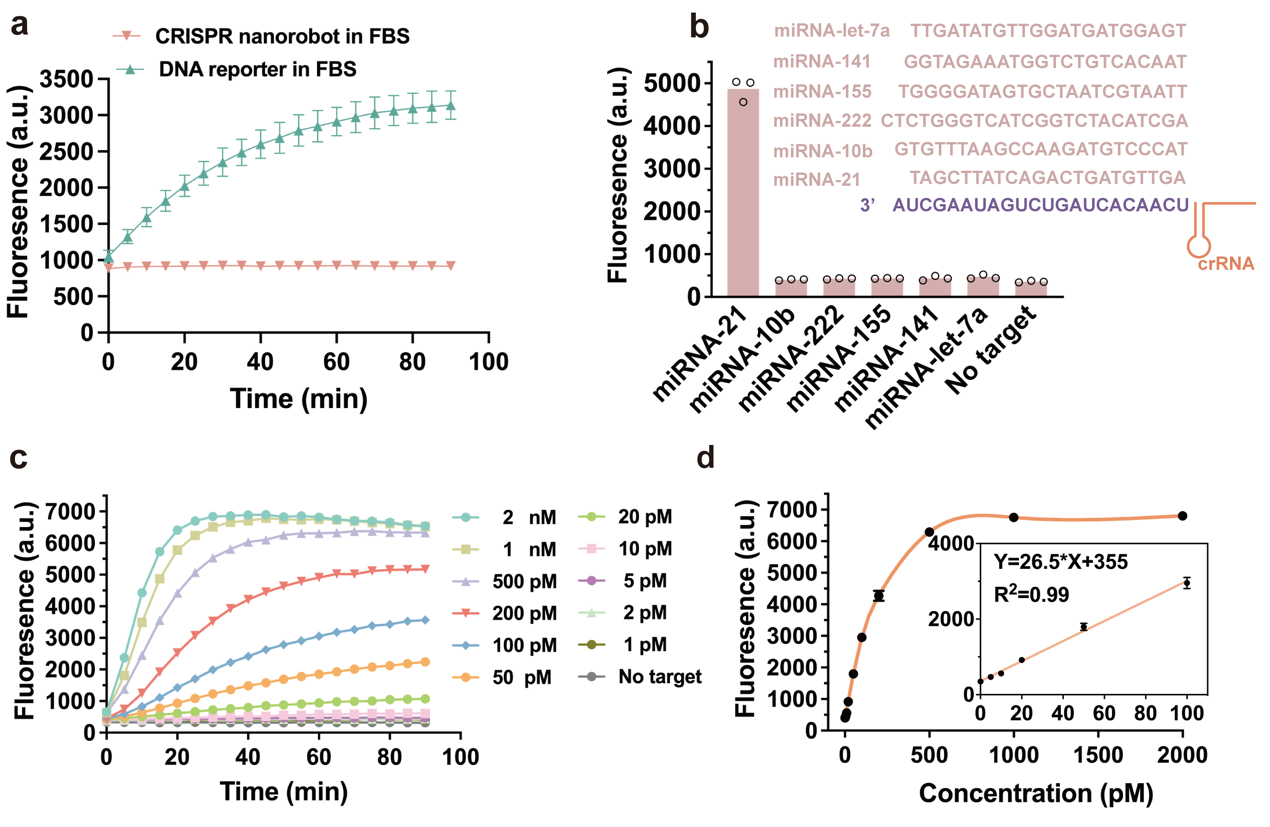
图4. CRISPR纳米机器用于RNA检测的稳定性、选择性和灵敏度。(a)在FBS存在下CRISPR纳米机器与常用DNA报告基因的稳定性的比较。DNA报告基因是一条短链DNA,两端用FAM和猝灭剂标记。DNA报告基因和CRISPR纳米机器分别与80% FBS混合。通过测量FAM的荧光强度随时间变化来表征DNA报告基因和CRISPR纳米机器的稳定性。(b)CRISPR纳米机器对miRNA-21靶标的选择性。只有miRNA-21靶标的序列与crRNA的序列互补。miR-let-7a、miR-141、miR-155、miR-222和miR-10 b的序列与crRNA不互补。(c)CRISPR纳米机器人响应不同浓度(0 - 2000 pM)的靶miRNA产生的荧光强度。(d)CRISPR纳米机器产生的荧光强度与miRNA浓度成线性关系。线性范围为1 - 100 pM(R2 = 0.99)。
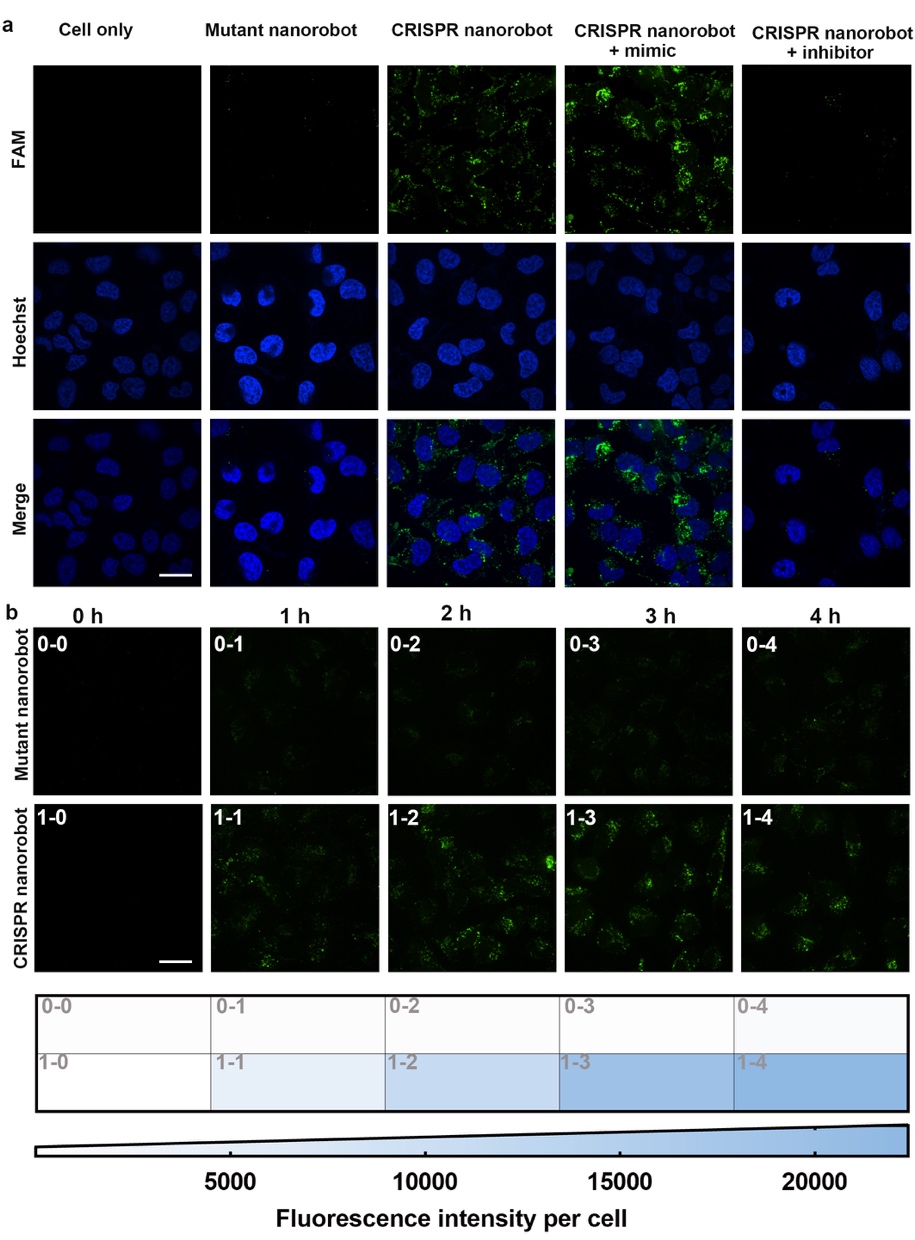
图5. CRISPR纳米机器的活细胞成像。(a)HeLa细胞在与CRISPR纳米机器或突变体纳米机器或没有纳米机器(阴性对照)孵育后的荧光图像。靶miRNA与CRISPR纳米机器的结合激活纳米机器并启动Cas12a的反式切割活性。活性Cas12a重复切割数百个锚定在AuNP表面上的FAM标记的底物链。这些FAM标记的底物片段从猝灭的AuNP中释放,产生放大的荧光。作为miRNA-21的模拟物的合成RNA序列用作阳性对照。使用miRNA-21抑制剂(与miRNA-21结合的互补DNA序列)作为额外的阴性对照。(b)在CRISPR纳米机器或突变体纳米机器的细胞内操作0、1、2、3和4小时后拍摄HeLa细胞的时程图像。每个图像中细胞的荧光强度被量化,从而允许评估CRISPR纳米机器的细胞内活性的时间动态。
该论文的第一作者为中国科学院生态环境研究中心2021届博士生袁爱姣,通讯作者为彭汉勇研究员。该团队还对核酸纳米机器设计、合成及应用等相关技术进行了系统综述(TrAC-Trend Anal Chem 2023,158, 116870.TrAC-Trend Anal Chem 2024,175, 117724.)。该工作得到了国家自然科学基金委面上项目、国家重点研发计划、中国科学院基础与交叉前沿科研先导专项(B类)等项目的支持。
